Các loài vi sinh vật mà chúng ta có thể nuôi cấy trong môi trường thạch trên đĩa chỉ là một phần rất nhỏ so với quần thể tồn tại trên thực tế. Vậy làm sao chúng ta có thể thu được những vi sinh vật đó? Kỹ thuật metagenomics có thể sẽ là câu trả lời.
“Con tôm ôm cây lúa” hay độc canh lúa sẽ mang lại hiệu quả cao hơn? Trong bối cảnh người nông dân đang ồ ạt chuyển sang mô hình luân canh tôm – lúa một cách tự phát, thậm chí là ‘quá độ’ hẳn sang chuyên tôm vì hiệu quả kinh tế cao hơn, rất nhiều nhà khoa học đã lăn xả tìm hiểu về tác động thực sự của những mô hình nuôi trồng này với mong muốn đưa ra các khuyến nghị phù hợp.
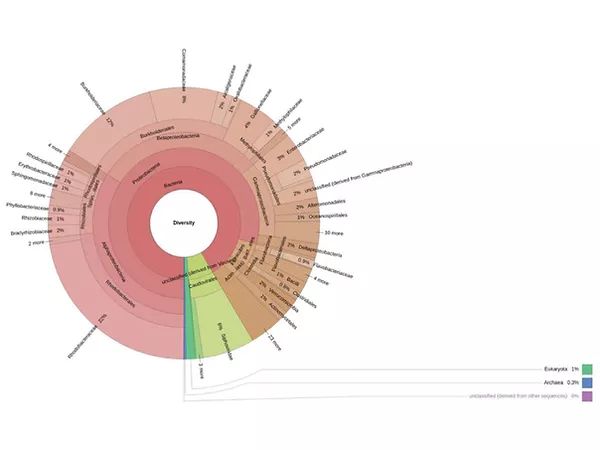
Bên cạnh những nghiên cứu về hiệu quả kinh tế, hay nghiên cứu về tác động của mô hình đến chất lượng đất về lâu dài, còn có sự hiện diện của một kỹ thuật mới có thể giúp phân tích, so sánh thành phần, độ đa dạng cả hệ vi sinh vật đất của hai mô hình canh tác lúa – lúa và luân canh tôm – lúa, đó là kỹ thuật metagenomics.
Thuật ngữ “metagenomics” được sử dụng lần đầu tiên trong một công bố của các nhà khoa học quốc tế vào năm 1998. Đây là ngành khoa học nghiên cứu về đa hệ gene (metagenome) – nguyên liệu di truyền được thu hồi trực tiếp từ các mẫu môi trường. “Theo cách truyền thống, chúng ta sẽ nuôi cấy vi sinh vật trong môi trường thạch trên đĩa petri, sau đó nhận diện hình thái khuẩn lạc, phân lập, giải trình tự, khai thác thông tin bộ gene để định danh vi sinh vật, xác định chức năng, đặc trưng của nó”, TS. Nguyễn Thị Mỹ Nương – Giảng viên Bộ môn Di truyền Đại học Khoa học Tự nhiên Đại học Quốc gia TP.HCM, chia sẻ tại buổi hội thảo “Giải trình tự Metagenomics – Phân tích hệ vi sinh môi trường” do Trung tâm Thông tin và Thống kê Khoa học và Công nghệ TP.HCM (CESTI) tổ chức vào tuần trước. Song, cần lưu ý rằng các loài vi sinh vật mà chúng ta có thể nuôi cấy trong môi trường thạch trên đĩa petri chỉ là một phần nhỏ so với số vi sinh vật tồn tại trên thực tế. Đa số chúng không tương thích với môi trường mà chúng ta cung cấp nên sẽ không phát triển trên đĩa petri. Tùy vào từng loại mẫu môi trường, số lượng vi sinh vật không nuôi cấy được dao động từ 99,0 đến 99,7%.
Trước hạn chế đó, metagenomics ra đời, cho phép chúng ta khai thác tối đa các gene của vi sinh vật không nuôi cấy được trong các quần thể sinh vật. Kỹ thuật này được ứng dụng nhiều trong nghiên cứu hệ vi sinh vật trong môi trường mà không cần phân lập, nuôi cấy những loài riêng lẻ. Có thể ứng dụng metagenomics trong nhiều mẫu môi trường đa dạng như đất, nước, biển, bùn, rễ cây, ruột, phân hay cơ thể động vật,…
“Về cơ bản, metagenomics trả lời hai câu hỏi: Vi sinh vật nào đang ở đó? Và chúng đang làm gì?”, TS. Nương đúc rút. Chị hiện là cố vấn của Công ty TNHH Khoa học KTest – chuyên cung cấp các giải pháp công nghệ tiên tiến trong lĩnh vực sinh học phân tử, di truyền và tin sinh học. Đây cũng là công ty đã áp dụng kỹ thuật metagenomics vào phân tích thành phần hệ sinh vật đất của hai mô hình canh tác tại Sóc Trăng.
Hợp tác với Đại học Cần Thơ, các nhà khoa học tại KTest đã xem xét DNA tổng số trong mẫu đất của hai mô hình canh tác được thu nhận; nhân bản trình tự gene 16S rRNA và giải trình tự trên hệ thống Illumina (16S metagenomics), so sánh dữ liệu gene thu nhận được với cơ sở dữ liệu để xác định thành phần vi sinh vật hiện diện. Theo đó, nhóm vi sinh chỉ thị ở hai mô hình canh tác lúa – lúa và lúa – tôm có tỷ lệ khác nhau: Firmicutes (28.6 và 62.9%), Actinobacteriota (22.4 và 9.5%), Chloroflexi (18.8 và 4.1%), Myxococcota (9.4 và 9.5%), và Acidobacteriota (4.4 và 0.6%). Như vậy, tỷ lệ hiện diện phyla (ngành) ở mô hình lúa – tôm đa dạng hơn mô hình thâm canh lúa.
Ngoài ra, KTest cũng tham gia vào nghiên cứu xác định nhanh thành phần và tỷ lệ hiện diện hệ vi sinh và xác định gene kháng kháng sinh trong nước ao tôm ở Ninh Thuận. Cụ thể, DNA tổng số trong nước ao tôm được thu nhận và giải trình tự shotgun metagenomics bằng thiết bị di động MinION (ONT platform), sau đó nhóm nghiên cứu xác nhận một số tác nhân gây bệnh bằng qPCR. Đáng chú ý, nhóm nhận thấy thành phần vi sinh nước ao tôm có 92% là vi khuẩn, 7% eukaryote, <1% là vi khuẩn cổ và virus, đặc biệt có sự hiện diện của WSSV phát hiện 0.01%, (Ct = 33); Vibrio parahaemolyticus: 0.03%. Nghiên cứu cũng xác định sự hiện diện của một số gene kháng kháng sinh nhóm tetracyline, beta lactam, sulfonamide, aminoglycoside.
Có thể nói, “tất cả các gene của vi sinh vật trong mẫu môi trường nếu được tập hợp lại sẽ là nguồn nguyên liệu vô cùng phong phú cho việc khai thác gene, cũng như tìm hiểu cơ chế tác động giữa các vi sinh vật đảm bảo sự ổn định, phát triển chung của hệ sinh thái”, như lời khẳng định của các nhà khoa học tại Viện Công nghệ Sinh học (Viện Hàn lâm KH&CN Việt Nam) và Đại học Sư phạm TP.HCM trong một bài báo vào năm 2015.
Thiết kế theo yêu cầu
“Bên cạnh những nghiên cứu kể trên, chúng tôi từng tiến hành một nghiên cứu xác định giới tính của chim theo nhu cầu của khách hàng. Có nhiều con chim rất nhỏ nên chủ nuôi không xác định được đó là chim trống hay chim mái để tiến hành lai giống cho phù hợp. Và chúng tôi đã làm được điều đó bằng kỹ thuật metagenomics – với chỉ một chiếc lông chim” – câu chuyện thoạt nghe ‘dở khóc dở cười’ của TS. Nguyễn Thị Mỹ Nương thực chất đã cho chúng ta thấy được điểm đặc biệt của KTest: quy trình phân tích metagenomics sẽ được công ty thiết kế dựa trên yêu cầu của khách hàng.
Theo đó, công ty sẽ thu thập hoặc nhận mẫu, sau đó sẽ tiến hành tách chiết DNA/RNA và chuẩn bị thư viện, kế đến là tiến hành giải trình tự và phân tích dữ liệu. Tùy thuộc vào yêu cầu cụ thể, khách hàng sẽ nhận được những kết quả phân tích cũng như dịch vụ khác nhau bao gồm: Kết quả phân tích thành phần các loài có trong mẫu với các chỉ tiêu như sự hiện diện và phân loại các loài, tỷ lệ các loài, độ da đạng…; kết quả so sánh thành phần loài ở các mẫu hay nhóm mẫu (nếu có nhiều mẫu); phân tích các gene chức năng (con đường biến dưỡng, gene kháng kháng sinh…) nếu phân tích Shotgun Metagenomics; báo cáo tóm tắt phương pháp, quy trình thực hiện; các kết quả phân tích khác theo yêu cầu khách hàng; tư vấn quy trình chuẩn bị mẫu, kết quả và tham gia công bố kết quả nếu khách hàng có yêu cầu.
Công ty KTest có hai cách tiếp cận khác nhau đối với kỹ thuật metagenomics. Cách thứ nhất, 16S Metabarcoding (cây phả hệ dựa trên rRNA) giúp xác định thành phần quần xã vi sinh vật dựa trên giải trình tự vùng gene chỉ thị như 16S rRNA (vi khuẩn), ITS (nấm),… nhằm tiết kiệm chi phí và nhanh chóng hiểu được cấu trúc của một cộng đồng vi sinh. Tuy nhiên, khả năng phân loại đến mức loài và hiểu về chức năng còn giới hạn và chỉ nhận diện nhóm vi sinh vật mục tiêu.
Ở cách tiếp cận thứ hai, thay vì chỉ giải trình tự gene chỉ thị, Shotgun Metagenomics giải trình tự toàn bộ vật liệu truyền trong mẫu môi trường. Điều này không chỉ cho phép xác định thành phần, tỷ lệ vi sinh vật hiện diện trong mẫu mà còn cung cấp thông tin chú giải thành phần các gene chức năng. Cách tiếp cận thứ hai này cần lượng dữ liệu và chi phí cao hơn cách tiếp cận thứ nhất.
Nhìn nhận những ưu điểm của kỹ thuật metagenomics, TS. Nguyễn Thị Mỹ Nương cho rằng kỹ thuật này có thể khỏa lấp những khoảng trống mà các kỹ thuật vi sinh vật truyền thống hay nuôi cấy như trước đây để lại. Do không phù hợp với các môi trường nuôi cấy đã có, thiếu các chất chuyển hóa hoặc phân tử tín hiệu do các thành viên trong cộng đồng tiết ra, rất nhiều vi sinh vật không thể nuôi cấy được. Bên cạnh đó, nuôi cấy theo phương pháp truyền thống chỉ nghiên cứu từng đối tượng riêng rẽ trong điều kiện phòng thí nghiệm, không nghiên cứu tổng thể mức độ phức tạp về phân loài của cộng đồng. Ngoài ra, việc nuôi cấy truyền thống cũng không nhận diện được các tương tác phức tạp giữa các thành viên trong cộng đồng với nhau.
Song chị vẫn lưu ý rằng metagenomics không phải là công cụ vạn năng có thể thay thế hoàn toàn phương pháp nuôi cấy truyền thống. Các nhà khoa học vẫn cần khắc phục thêm một số hạn chế của metagenomics như không thể tách chiết hoàn toàn ADN của vi sinh vật trong môi trường, quá trình giải trình tự có thể bỏ sót các vi sinh vật có độ phong phú thấp, và không có “tiêu chuẩn vàng” cho phần mềm xử lý trình tự.
Dẫu vậy, cho đến hiện tại, “metagenomics là công cụ hữu ích với rất nhiều tiềm năng chưa được khai phá hết. Các vấn đề từ kháng kháng sinh, bệnh nhiễm, năng suất trồng trọt, chăn nuôi, ô nhiễm môi trường và biến đổi khí hậu đều có liên quan đến biến đổi quần xã vi sinh vật. Chính vì vậy, hiểu về thế giới của vi sinh vật, chúng ta có thể mở ra tiềm năng của chúng để cải thiện cuộc sống của chính chúng ta”, TS. Nương chia sẻ. Gần đây, công ty KTest đã tiến hành thành công nghiên cứu phân tích thành phần vi khuẩn trong mẫu sữa từ bò bệnh viêm vú; nghiên cứu xác định tác nhân gây bệnh ở mèo bệnh chưa rõ nguyên nhân bằng metagenomics.
“Bản thân các nhà cố vấn của KTest đều là những nhà khoa học thuộc Đại học Khoa học Tự nhiên (ĐHQG-TP.HCM). Chúng tôi hiểu cảm giác khó khăn của các nhà nghiên cứu khi các công ty có trang bị hệ thống giải trình tự thế hệ mới rất ít ỏi. Chúng tôi cũng từng gửi mẫu ra nước ngoài, nhưng việc hệ thống vận chuyển trục trặc, mẫu bị hư hại khi chuyển đến v.v. là những việc khó tránh khỏi”, chị nhớ lại. “Vì vậy, chúng tôi còn hướng đến xây dựng công ty như một nhà cung cấp dịch vụ giải trình tự DNA thế hệ mới hiệu quả trong nước, từ đó có thể hỗ trợ các nhà khoa học trong lĩnh vực này tiến hành công trình của mình một cách dễ dàng hơn”.
Metagenomics không phải là công cụ vạn năng có thể thay thế hoàn toàn phương pháp nuôi cấy truyền thống. Các nhà khoa học vẫn cần khắc phục thêm một số hạn chế của metagenomics như không thể tách chiết hoàn toàn ADN của vi sinh vật trong môi trường, quá trình giải trình tự có thể bỏ sót các vi sinh vật có độ phong phú thấp, và không có “tiêu chuẩn vàng” cho phần mềm xử lý trình tự.
TS. Nguyễn Thị Mỹ Nương
Nguồn: Khoa học phát triển
- Sản lượng tôm tăng mạnh vào tháng đầu năm, xuất khẩu hưởng lợi từ nhu cầu dịp Tết
- Quảng Trị: Nuôi tôm trong bể nổi xi măng, giải pháp nuôi trồng bền vững
- Giá tôm liên tục dao động dịp cận Tết
- Lâm Đồng đặt mục tiêu trở thành trung tâm cung ứng giống tôm hàng đầu cả nước
- Vị thế cạnh tranh của tôm Việt Nam tại thị trường Trung Quốc
- Cần Thơ: Khuyến cáo kỹ thuật nuôi tôm nước lợ thích ứng thời tiết năm 2026
- An Giang: Vuông tôm Miệt Thứ vào vụ
- Nhiều tầng rào cản tại Mỹ bào mòn sức cạnh tranh của tôm Việt
- Thanh Hóa: Tiên phong ứng dụng công nghệ cao, làm giàu từ nuôi tôm
- Nuôi tôm công nghệ cao: Động lực tăng trưởng mới cho Vĩnh Long
Tin mới nhất
CN,08/02/2026
- Sản lượng tôm tăng mạnh vào tháng đầu năm, xuất khẩu hưởng lợi từ nhu cầu dịp Tết
- Quảng Trị: Nuôi tôm trong bể nổi xi măng, giải pháp nuôi trồng bền vững
- Giá tôm liên tục dao động dịp cận Tết
- Lâm Đồng đặt mục tiêu trở thành trung tâm cung ứng giống tôm hàng đầu cả nước
- Vị thế cạnh tranh của tôm Việt Nam tại thị trường Trung Quốc
- Cần Thơ: Khuyến cáo kỹ thuật nuôi tôm nước lợ thích ứng thời tiết năm 2026
- An Giang: Vuông tôm Miệt Thứ vào vụ
- Nhiều tầng rào cản tại Mỹ bào mòn sức cạnh tranh của tôm Việt
- Thanh Hóa: Tiên phong ứng dụng công nghệ cao, làm giàu từ nuôi tôm
- Nuôi tôm công nghệ cao: Động lực tăng trưởng mới cho Vĩnh Long
- Con tôm sú thế hệ mới là thứ tôm gì mà ở Cần Thơ “nghe tên mà ham”, nhà nào nuôi là trúng?
- Ngành tôm miền Bắc: Nuôi bài bản, đẩy lùi rào cản
- Nông dân đổi đời nhờ mô hình canh tác hiện đại
- Trí tuệ nhân tạo (AI): Mở lối nghề nuôi trồng thuỷ sản thông minh
- Thực tiễn sản xuất tôm: Câu chuyện về giống nội địa và ngoại nhập
- Chứng nhận ASC: Cơ hội nâng tầm thuỷ sản Việt Nam
- “Đôi bạn cùng tiến” có lợi cho nuôi tôm?
- Làm giàu từ nuôi tôm công nghệ cao
- Bình Định tăng cường quản lý hoạt động khai thác, bảo vệ nguồn lợi thủy sản
- Thủy sản Việt Nam tìm cách thoát ‘bẫy phụ thuộc’
- Xuất khẩu tôm: Cần xây dựng thương hiệu gắn với chất lượng
- Giá trị xuất khẩu thủy sản khởi sắc những tháng đầu năm
- Tăng cường kiểm soát thủy sản Việt Nam xuất khẩu vào châu Âu
- Đề xuất thành lập “nhóm đặc nhiệm” về thương mại nông sản Việt Nam – Singapore
- Hà Nội thúc đẩy phát triển nguồn lợi thủy sản
- Sản lượng thủy sản tháng đầu năm tăng nhẹ so với cùng kỳ năm ngoái
Các ấn phẩm đã xuất bản
- Giá tôm xuất khẩu tăng mạnh, tạo động lực phục hồi sản xuất và nâng tầm giá trị ngành tôm
- Gần 500 hộ nuôi khẳng định hiệu quả Advance Pro – Tiến bộ kỹ thuật của Grobest
- Người nuôi tôm phập phồng với “ngày nắng, đêm mưa”
- Động lực phát triển đột phá ngành thủy sản
- Công nghệ sinh học toàn diện: Giải pháp nuôi tôm thành công từ Tâm Việt
- Biện pháp kiểm soát khí độc Nitrite (NO2) trong mô hình nuôi tôm TLSS-547
- Ngành chức năng và nông dân Quảng Nam cùng gỡ khó cho nuôi tôm nước lợ
- Thời tiết bất lợi gây thiệt hại hơn 27 triệu con tôm sú, tôm thẻ nuôi
- [Tuyển dụng] – Công ty TNHH Seven Hills Trading tuyển dụng nhiều vị trí hấp dẫn trong năm 2025
- Nhiều diện tích tôm nuôi của Nghệ An bị bệnh đốm trắng
- An toàn sinh học: Giải pháp then chốt cho bài toán dịch bệnh thủy sản
- Sử dụng sóng siêu âm để tính sinh khối ao nuôi tôm
- Máy sưởi ngâm: Cách mạng hóa nghề nuôi tôm ở Việt Nam
- Waterco: Giải pháp thiết bị hàng đầu trong nuôi trồng thủy sản
- GROSHIELD: “Trợ thủ đắc lực” giúp tôm đề kháng vững vàng hàng ngày, sẵn sàng về đích
- Năm mới, nỗi lo cũ: “Làm sao để tăng cường đề kháng cho tôm?”
- Vi sinh: Giải pháp mục tiêu toàn diện
- Grobest Việt Nam: Tiên phong ra mắt sản phẩm thức ăn chức năng hàng ngày Groshield, nâng cao tối đa sức đề kháng, hướng đến những vụ tôm về đích thành công trong năm tới
- Solagron Vietnam: Nhà sản xuất vi tảo công nghiệp đầu tiên mang dấu ấn Việt Nam
- Giải pháp giảm phát thải trong nuôi trồng thủy sản từ bột cá thủy phân
















































